TECHOZYME
QUANTIFICATION ET ANALYSE DE LA PURETÉ DES ACIDES NUCLÉIQUES PAR SPECTROPHOTOMÉTRIE DSDNA, SSDNA, RNA, OLIGONUCLÉOTIDES
En biologie moléculaire, il est important de quantifier et d’analyser la pureté des acides nucléiques après leur purification. La méthode la plus répandue pour le dosage d’acides nucléiques est la spectrophotométrie qui mesure l’absorbance (ou densité optique) des acides nucléiques à 260 nm (absorbent dans l’ultraviolet). Parallèlement on détermine leur pureté en mesurant l’absorbance à 280 nm, 230 nm et 320 nm.
Ce TechOzyme, passe en revue le principe du dosage en spectrophotométrie. En complément, une brève présentation est faite sur la quantification en fluorescence.
SPECTROPHOTOMÉTRIE D’ABSORPTION : LOI DE BEER-LAMBERT
—————
La spectrophotométrie d’absorption dans l’UV est une méthode très courante en laboratoire. Elle repose sur la propriété des molécules (Ex. acides nucléiques, protéines …) d’absorber des radiations lumineuses à une longueur d’onde déterminée.
L’absorbance (A) aussi appelée Densité Optique (D.O) est la mesure quantitative d’absorption d’une radiation UV-visible à une longueur d’onde donnée par un composé ou un échantillon :
A = log (I0/I)
. I0 = intensité de la lumière incidente
. I = intensité transmisse après le passage à travers la cuve contenant la solution ou l’échantillon
Bouguer, Lambert et Beer ont étudié les relations qui existent entre I0 et I et déterminé que l'intensité d'une lumière monochromatique qui traverse et qui est absorbée par une solution, décroît de façon exponentielle :
I = I0e-klc
. l = distance traversée par la lumière (largeur de la cuve) (en cm) ou longueur du trajet optique
. C = concentration des substances absorbantes
. k = constante caractéristique de la substance étudiée
Cette équation peut se réécrire en :
A = log (I0/I) = klC/2,3 = ε.l.c
ε est le coefficient d'extinction molaire ; spécifique de la substance étudiée à une longueur d'onde donnée.
« C » est la molarité en mol.l-1, « ε » est en L.mol-1.cm-1, et « l » en cm, on obtient la relation de la loi de Beer-Lambert:
A = ε.l.c
La loi de Beer-Lambert s'applique aux radiations monochromatiques et aux solutions diluées ou en faible concentration (c<0,1 mol.l-1) car dans ce cas les propriétés des molécules (association, complexation …) ne sont pas modifiées de manière significative.
Le spectre d’absorption UV-visible décrit la variation de l’absorbance (A) en fonction de la longueur d’onde l. Le coefficient d’extinction ε pour un composé donné varie en fonction des longueurs d’onde. Un composé est caractérisé par sa longueur d’onde d’absorption maximale (λmax), son coefficient d’extinction à la λmax (εmax) et son spectre d’absorption.
ANALYSE QUANTITATIVE : MESURE DE LA CONCENTRATION
—————
Les acides nucléiques absorbent dans l’UV à λmax = 260 nm. En mesurant l’absorbance d’une solution d’acide nucléique pure à 260 nm, on obtient sa concentration à l’aide de l’équation de la loi Beer-Lambert. En pratique, c’est une équation modifiée qui est employée :
cm = (A.e) / l
. cm = concentration massique d’acide nucléique en ng/µl
. A = absorbance à 260 nm
. e = coefficient d’extinction massique ou la constante en ng.cm/µl
. l = trajet optique en cm
Le coefficient d’extinction ε dépend de la séquence d’ADN ou d’ARN et est spécifique à chaque ADN/ARN. Ci-dessous à titre indicatif, les coefficients d’extinction des différents nucléotides :
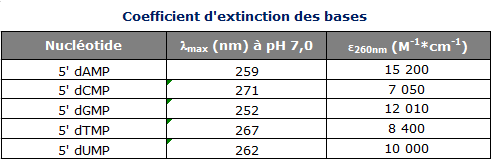
En pratique, pour chaque séquence d’ADN/ARN, il n’est pas nécessaire de calculer le coefficient d’extinction (ε) spécifique. Des coefficients d’extinction estimatifs, acceptés par la communauté scientifique, sont généralement utilisés pour calculer les concentrations des acides nucléiques et qui sont suffisamment précises pour les expériences de biologie moléculaire. Ces coefficients d’extinction sont listés ci-dessous :
. ADN double brin = ~50 ng.cm/µl
. ADN simple brin = ~33 ng.cm/µl
. ARN = ~40 ng.cm/µl
Une exception existe pour les oligonucléotides qui sont en simple brin et dont les séquences sont très courtes. La composition de séquence des oligonucléotides influence significativement l’absorbance contrairement aux ADN/ARN simple brin ou double brin plus longs.
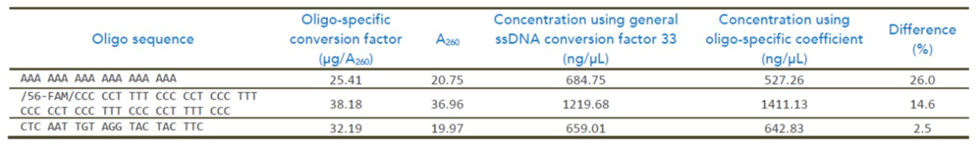
La formule ci-dessous est recommandée pour calculer les concentrations des oligonucléotides :
C (pmol/µl) = A260 x 100(1,5NA + 0,71NC + 1,20NG + 0,84NT)
Il n’est pas rare d’observer des différences de 5 à 10% parmi les résultats de quantification des oligonucléotides faute d’utiliser un coefficient d’extinction correct. Un exemple de ces variations est donné dans le tableau ci-dessous :
Quand le trajet optique et le coefficient d’extinction sont connus, il possible de calculer les facteurs de conversion entre l’absorbance et la concentration. Dans le cas où une cuvette de 1 cm de largeur est utilisée, on obtient les facteurs de conversion suivants :
. 1 unité A260nm = ~50 µg/ml d’ADN double brin
. 1 unité A260nm = ~33 µg/ml d’ADN simple brin
. 1 unité A260nm = ~40 µg/ml d’ARN simple brin
Pour la méthode de spectrophotométrie d’absorption, la concentration d’acide nucléique minimale détectable (limite de détection) par la spectrophotométrie d’absorption dépend du trajet optique. Plus le trajet optique est long, plus la limite est faible. Par exemple, pour un trajet optique de 1 mm, la limite de détection est de 750 ng/µl et elle est 10 fois plus faible quand le trajet optique est 10 fois plus long. L'avantage d'un trajet optique faible est qu'il est possible de détecter des concentrations élevées sans diluer.
ANALYSE DE LA PURETÉ
—————
Plusieurs paramètres basés sur les absorptions spectrophotométriques sont utilisés pour analyser la pureté d’une solution d’acides nucléiques.
Le ratio A260 nm/280 nm
Initialement en 1942, Warburg et Christian (1) avaient publié un rapport expliquant l’intérêt de l’utilisation du ratio A260 nm/280 nm pour détecter la contamination d’acides nucléiques présente dans une solution de protéines. Maintenant, ce ratio est plus souvent utilisé pour évaluer la contamination de protéines dans une solution d’acides nucléiques.
Voici un exemple de ce ratio de l’ADN de thymus de veau pur dans 1X de tampon TNE à 25 µg/ml
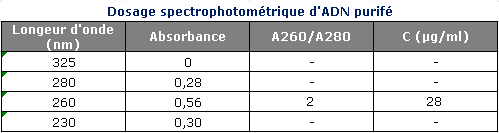
D’une manière générale, la pureté d’une solution d’acide nucléique est considérée comme acceptable lorsque le ratio A260 nm/A280 nm est compris entre 1,8 – 2,0 pour l’ADN et entre 2,0 – 2,2 pour l’ARN. Même si les études menées par Glasel montrent qu’il y a 60% de protéines et 40% d’acides nucléiques lorsque ce ratio est égal à 1,81 (2).
Comme ce ratio varie significativement en fonction du pH et de la force ionique (3), il est important de prendre en compte ces paramètres pour interpréter ce ratio.
Par exemple, le ratio d’une solution plus acide est de 0,2 – 0,3 plus faible et il est de 0,2 – 0,3 plus élevé quand la solution est plus basique. Enfin, le ratio A260 nm/A280 nm varie d’un ADN/ARN à l’autre, car il dépend de leur composition de séquences. Voici les ratios A260 nm/A280 nm mesurés pour chaque nucléotide :
. Guanine : 1,15
. Adénine : 4,50
. Cytosine : 1,51
. Uracile : 4,00
. Thymine : 1,47
Le ratio A260 nm/A230 nm
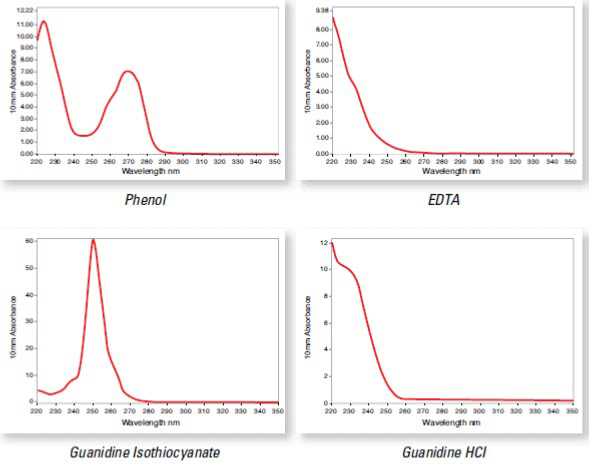
Le ratio A260/A230 est un deuxième indicateur de pureté, il est généralement plus élevé que le ratio A260/A280. Un ratio A260/A230 compris entre 2,0 – 2,2 indique une bonne pureté des acides nucléiques. Lorsque ce ratio est significativement plus faible que 2,0 – 2,2, cela révéle la présence de contaminants absorbant à 230 nm dans la solution. Plusieurs contaminants absorbant à 230 nm proviennent de l’échantillon ou de la purification, comme par exemple le phénol, l’isothiocyanate de guanidine, les carbohydrates, les peptides, l’acide humique, l’urée, l’EDTA, les polysaccharides …. Leur présence est détectable grâce à ce ratio.
Ci-dessous à titre d’exemple, les spectres du phénol, de l’EDTA, de la guanidine isothiocyanate et de la guanidine HCL.
Le spectre d’absorption
Le spectre d’absorption d’une solution d’acides nucléiques pure est représenté par une courbe caractéristique représenté par la figure 1. Un changement de profil de cette courbe est un bon indicateur de l’impureté d’une solution y compris pour les solutions d’acides nucléiques.
La figure 2 montre des décalages des courbes de spectre d’absorption d’un même ADN en solution pure ou contenant des contaminants.
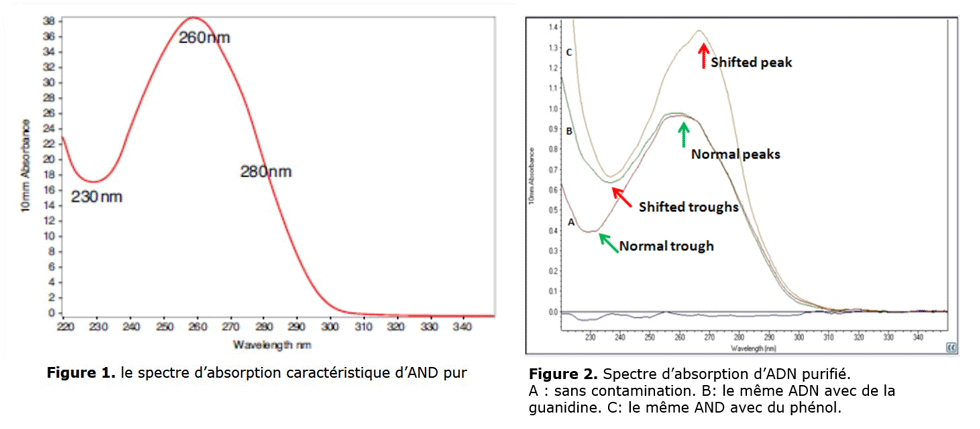
L’absorbance à A320 nm
L’absorbance à 320 nm est un indicateur de la présence de particules dans la solution et/ou d’impuretés présentes sur la paroi de la cuvette ou à la surface de mesure (sur le piédestal).
Le cas des oligonucléotides
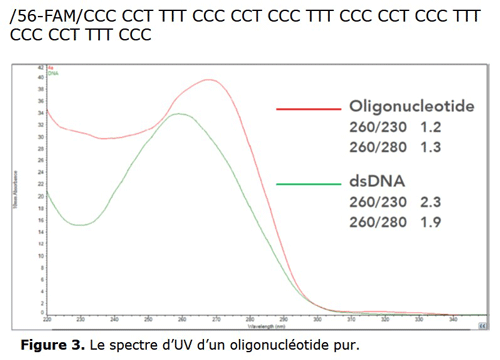
Le spectre et les ratios A260/280, A260/230 pour un oligonucléotide ne sont pas les mêmes que pour un ADN. La figure 3 compare les spectres d’absorption d’un oligonucléotide pur et d’un ADN double brin pur. Il est important de noter que la lmax pour chaque nucléotide (A, T, C, G) est différent et spécifique (comprises entre 255 – 280 nm) et que chaque nucléotide contribue au spectre d’absorption. Etant donné que les oligonucléotides sont en simple brin et très courts, leur spectre d’absorption varie significativement en fonction de leur séquence. Le spectre et les ratios A260/280 et A260/230 des oligonucléotides sont décalés par rapport à l’ADN et donc les ratios de pureté de référence pour les ADN ne sont pas applicables aux oligonucléotides.
Par exemple, la figure 3 montre que l’oligonucléotide pur n’a pas les mêmes ratios A260/280 et A260/230 que ceux de l’ADN double brin pur.
Les facteurs qui influencent les ratios de pureté :
Les facteurs comme le pH, la force ionique de la solution d’acides nucléiques (3), la présence de bulles d’air, les fluorochromes (pour les oligonucléotides marqués) et également la précision du spectrophotomètre impactent les ratios de pureté. Selon les résultats publiés par Wilfinger en 1997, le ratio A260/A280 d’un ARN pur dans une solution plus acide est significativement plus faible, pouvant induire en erreur sur l’interprétation de la pureté. Il est donc important d’utiliser le même tampon pour le blanc et pour la solution d’acides nucléiques lors du dosage par le spectrophotomètre. Un léger décalage de la précision de longueur d'onde d’un spectrophotomètre peut modifier significativement les ratios A260/A280. Par exemple, un décalage de +/- 1 nm de précision de longueur d'onde se traduira par une variation de +/- 0,2 pour le ratio A260/A280. Comme beaucoup de spectrophotomètres réclament une précision de 1 nm, il est possible d’observer une différence jusqu’à 0,4 pour les ratios A260/A280 d’un même échantillon d'acides nucléiques obtenus avec deux spectrophotomètres différents. Il est donc important de vérifier régulièrement la performance et la précision de son spectrophotomètre.
QUANTIFICATION PAR FLUORESCENCE (FLUOROMÉTRIE)
—————
La quantification par fluorescence est une méthode plus sensible que la spectrophotométrie qui permet d’obtenir une sensibilité de l’ordre du pg/µl. De plus, elle est plus spécifique et n’est pas impactée par la présence de contaminants dans la solution d’acides nucléiques. Elle est particulièrement intéressante lorsque la solution contient à la fois de l’ADN et de l’ARN, car elle permet de les quantifier spécifiquement. Mais contrairement à la spectrophotométrie, la fluorimétrie ne permet pas d’analyser la pureté et ni d’identifier des contaminants dans une solution d’acides nucléiques.
APPLICATIONS DE LA SPECTROPHOTOMÉTRIE
—————
La spectrophotométrie est utilisée au quotidien dans les laboratoires de biologie moléculaire pour les dosages d’ADN, d’ARN et des oligonucléotides après leur purification. C’est une méthode simple, rapide et précise. Elle est également utilisée pour le NGS et l’identification de contaminants chez les plantes :
NGS
Simbolo et al (4) compare trois méthodes pour la quantification d’ADN extraits à partir d’échantillons d’histopathologie (FFPE):
. Le Qubit™ (un fluorimètre associé avec des réactifs spécifiques pour la quantification spécifique d’ADN ou d’ARN)
. La qPCR
L’objectif est de valider la qualité et la quantité de ces ADN après leur purification pour la construction de banque NGS (Next Generation Sequencing). Les auteurs recommandent d’utiliser en parallèle le NanoDrop® et le Qubit™ (moins coûteux) ou le NanoDrop® et la qPCR (plus couteux), car ni le Qubit™ ni la qPCR ne permet la détection de contaminants présents dans les ADN purifiés. Les ratios A260/A230 obtenus par le NanoDrop® sont de bons indicateurs de pureté.
La détection de la contamination de polysaccharides
Les échantillons de plantes sont souvent riches en polysaccharides, un inhibiteur de la PCR. Les ADN extraits à partir des plantes en contiennent souvent. Comme les polysaccharides absorbent à 230 nm, le ratio A260/A230 est sélectionné pour estimer la contamination en polysaccharides de ces ADN.
QUESTIONS FRÉQUEMMENT POSÉES
—————
Pourquoi la concentration de mon échantillon est-elle négative ?
Ceci peut être dû à l’utilisation d’une solution inappropriée pour effectuer le « blanc » ou bien le « blanc » a été effectué dans une cuvette sale ou sur une surface de piédestal sale. Il est important d’utiliser pour le « blanc » une solution dont la composition et le pH sont les mêmes que ceux utilisés pour les acides nucléiques purifiés.
Mes résultats ne sont pas reproductibles
Votre solution d’acides nucléiques n’est peut-être pas homogène. Les ADN de grandes tailles comme les ADN génomiques peuvent être plus difficiles à dissoudre. Il faut bien mélanger à l’aide d’un vortex la solution contenant ces ADN.
Ou bien, les concentrations d’acides nucléiques sont trop faibles ou bien elles sont trop élevées par rapport aux limites de quantification du spectrophotométre, les mesures ne sont donc pas reproductibles.
Voici les limites de détection des ADN double brins pour les NanoDrop® :
NanoDrop ONE : 2 à 27 500 ng/µl
NanoDrop ONEc : 0,2 à 75 ng/µl en mode cuvette ou 2 à 27 500 ng/µl en mode piédestal
NanoDrop 2000 : 2 à 15 000 ng/µl
NanoDrop 2000c : 0,4 à 75 ng/µl en mode cuvette ou 2 à 15 000 ng/µl en mode piédestal
NanoDrop 8000 : 2,5 à 3750 ng/µl
NanoDrop 1000 : 2 à 3750 ng/µl
La spectrophotométrie permet-elle de quantifier les acides nucléiques marqués par des fluorochromes ?
Oui. Dans ce cas, il est important de prendre en compte l’absorbance à 260 nm des fluorochromes. Le spectrophotométre NanoDrop® inclut le mode de mesure « Microarray » qui permet de mesurer simultanément les absorbances des acides nucléiques et des fluorochromes et de calculer la concentration de l’acide nucléique.
LE SAVIEZ-VOUS ?
—————
Le ratio A260/A280 d’une série de dilution n’est pas toujours constant
Même si la dilution est parfaite, le ratio A260/A280 peut varier à cause du changement de pH suite aux dilutions.
La spectrophotométrie ne permet pas de discriminer les ADN et les ARN dans une même solution
L’absorbance d’une solution est le résultat des absorbances de l’ensemble des acides nucléiques présents. Ainsi, si une solution d’ADN contient de l’ARN ou si des contaminants absorbent à 260 nm, la concentration de l’ADN sera surestimée. Si une solution contient de l’ADN dégradé et non dégradé, la concentration mesurée par la spectrophotométrie correspond aux deux types d’ADN. Il est donc important de traiter les ADN par de la RNase ou les ARN par de la DNase avant la quantification par spectrophotométrie.
RÉFÉRENCES BIBLIOGRAPHIQUES
—————
1.Warburg, O. and W. Christian. 1942. Isolation and crystallization of enolase. Biochem. Z. 310:384-421
2.Jay A. Glasel, 1995. Validity of nucleic acid purities monitored by 260nm/280nm absorbance ratios. BioTechniques, 18(1):62-3.
3.William W. Wilfinger, Karol Mackey, and Piotr Chomczynski. 1997. Effect of pH and Ionic Strength on th spectrophotometric Assessment of Nucleic Acid Purity. BioTechniques, 22:474-481
4.Michele Simbolo et al. 2013. DNA qualification workflow for next generation sequencing of histopathological samples. Plos One, 8(6):e62692


